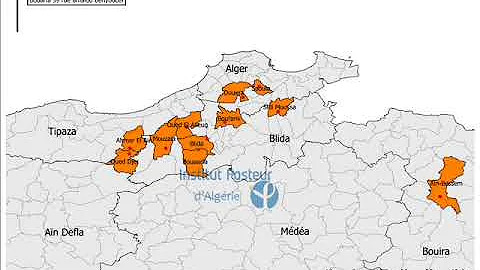
Premières analyses de séquences des souches algériennes de SARS-CoV-2
Afin de déterminer la relation entre les séquences algériennes et les variantes circulantes du SARS-CoV-2/hCOV-19, un arbre évolutif a été déduit en utilisant tous les génomes humains complets disponibles auprès du GISAID GISAID (base de données à l’origine des séquences de la grippe ; Global Initiative on Sharing All Influenza Data) au 20 mars 2020, ainsi que les nouvelles séquences soumises (en attente sur le GISAID). Un alignement de séquences multiples de 691 génomes complets a été généré et a été conservé manuellement. Un arbre de maximum de vraisemblance (ML) a été estimé en utilisant TreeTime tel qu’implémenter dans Nextstrain.
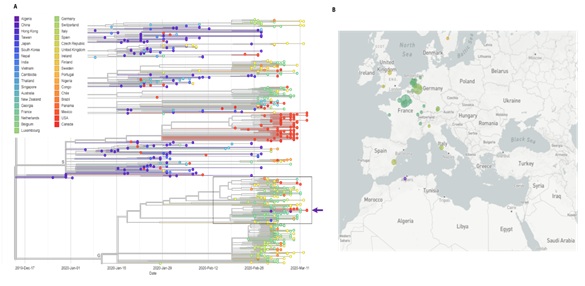
Figure 1.
- Estimation de l'arbre phylogénétique à probabilité maximale pour des séquences
Complètes du génome de 691 souches de virus COVID-19. Les données sur le génome utilisées.
Ici en plus des séquences nouvellement générées ont été mises à disposition dans la plateforme.
GISAID, à partir du 20 mars 2020. La flèche violette indique la position des séquences Algériennes et le détail du carré noir est montré dans la Fig.2.
- Visualisation des séquences: SARS-CoV-2 disponibles autour de la mer Méditerranée
Deux génomes complets ont été obtenus à partir d'échantillons des souches de l’Institut Pasteur d'Algérie. Ces séquences diffèrent par 3 mutations ponctuelles et s'inscrivent dans le clade G tel que défini par le GISAID (Figure 1, flèche violette). D’autres sont en cours.
NB/Les séquences sont disponibles et publiées dans la plateforme GISAID.
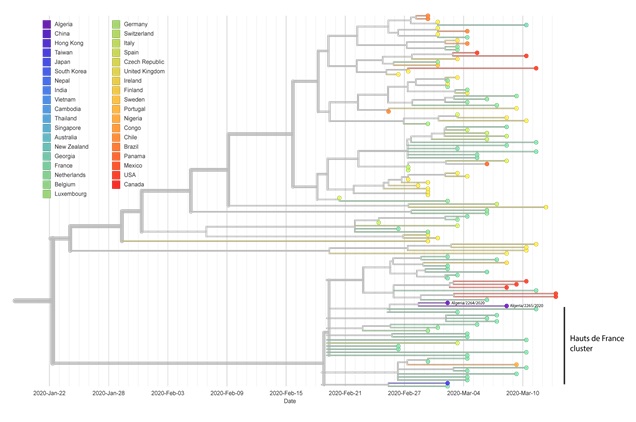
Figure 2. Vue agrandie de la section de la phylogénie comprenant les deux premières séquences algériennes.
La figure 2 montre plus en détail la position des deux premières séquences algériennes (en violet), qui se ramifient au sein d'un cluster français en cours (en vert), ce qui suggère une introduction unique du SARS-CoV-2 en Algérie, peut-être à partir de la France, en attendant d'autres données sur les séquences.